Imitar as condições fisiológicas ajuda os pesquisadores a encontrar ligantes metálicos
Os pesquisadores desenvolveram um método para identificar pequenas moléculas que se ligam a íons metálicos. Os íons metálicos são essenciais na biologia. Mas identificar com quais moléculas – e especialmente com quais moléculas pequenas – esses íons metálicos interagem pode ser um desafio.
Para separar metabólitos para análise, os métodos metabolômicos convencionais usam solventes orgânicos e pHs baixos, que podem causar a dissociação de complexos metálicos. Pieter C. Dorrestein, da Universidade da Califórnia em San Diego, e colegas de trabalho queriam manter os complexos juntos para análise, imitando as condições nativas encontradas nas células. Mas se usassem condições fisiológicas durante a separação das moléculas, teriam que reotimizar as condições de separação para cada condição fisiológica que quisessem testar.
Em vez disso, os pesquisadores desenvolveram uma abordagem em dois estágios que introduz condições fisiológicas entre uma separação cromatográfica convencional e uma análise espectrométrica de massa (Nat. Chem. 2021, DOI: 10.1038/s41557-021-00803-1). Primeiro, eles separaram um extrato biológico usando cromatografia líquida convencional de alta eficiência. Em seguida, ajustaram o pH do fluxo que sai da coluna cromatográfica para imitar as condições fisiológicas, adicionaram íons metálicos e analisaram a mistura com espectrometria de massa. Eles executaram a análise duas vezes para obter espectros de massa de pequenas moléculas com e sem metais. Para identificar quais moléculas se ligam aos metais, eles usaram um método computacional que utiliza formas de pico para inferir conexões entre os espectros das versões ligadas e não ligadas.
Uma maneira de imitar ainda mais as condições fisiológicas, diz Dorrestein, seria adicionar altas concentrações de íons como sódio ou potássio e baixas concentrações do metal de interesse. “Torna-se um experimento de competição. Basicamente, ele dirá: OK, essa molécula nessas condições tem mais propensão a se ligar ao sódio e ao potássio ou a esse metal único que você adicionou”, diz Dorrestein. “Podemos infundir muitos metais diferentes simultaneamente e podemos realmente compreender a preferência e a seletividade nesse contexto.”
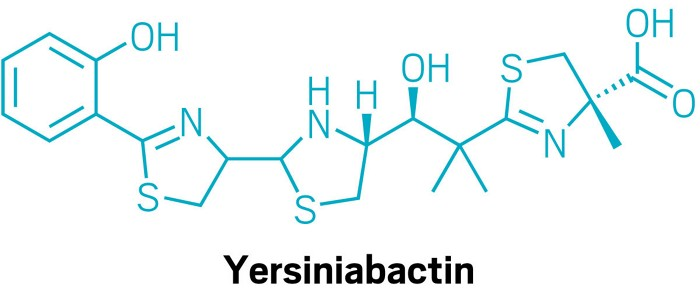
Em extratos de cultura de Escherichia coli, os pesquisadores identificaram compostos conhecidos de ligação ao ferro, como yersiniabactina e aerobactina. No caso da yersiniabactina, descobriram que ela também pode ligar-se ao zinco.
Os pesquisadores identificaram compostos ligantes de metais em amostras tão complexas quanto a matéria orgânica dissolvida do oceano. “Essa é absolutamente uma das amostras mais complexas que já observei”, diz Dorrestein. “É provavelmente tão complexo quanto, se não mais complexo, que o petróleo bruto.” O método identificou o ácido domóico como uma molécula de ligação ao cobre e sugeriu que ele se liga ao Cu2+ como um dímero.
“Uma abordagem ômica para identificar todos os metabólitos de ligação a metais em uma amostra é extremamente útil devido à importância da quelação biológica de metais”, escreve Oliver Baars, que estuda metabólitos de ligação a metais produzidos por plantas e micróbios na Universidade Estadual da Carolina do Norte. e-mail.
“Dorrestein e colegas de trabalho fornecem um ensaio elegante e muito necessário para investigar melhor qual poderia ser o papel fisiológico dos íons metálicos na célula”, escreve Albert JR Heck, pioneiro em análises de espectrometria de massa nativa na Universidade de Utrecht, por e-mail. “Um possível próximo passo seria extrair os metabólitos da célula em condições nativas e fracioná-los também em condições nativas, para ver quais metabólitos carregam quais íons metálicos celulares endógenos.”
Notícias de Química e Engenharia
ISSN0009-2347
Direitos autorais © 2021 Sociedade Química Americana
Horário da postagem: 23 de dezembro de 2021

